搜索网站、位置和人员


搜索网站、位置和人员


个人简介
史竹兵,2016年获同济大学博士学位。2010-2018年,在中国科学院上海生命科学研究院生物化学与细胞生物学研究所从事癌症与免疫相关大分子复合物的结构与功能研究。2018-2021年,在美国得克萨斯大学西南医学中心于洪涛教授实验室进行博士后研究,重点探索染色体折叠的分子机制。2021年9月,加入西湖大学生命科学学院,担任特聘研究员,博士生导师。
学术成果及研究方向
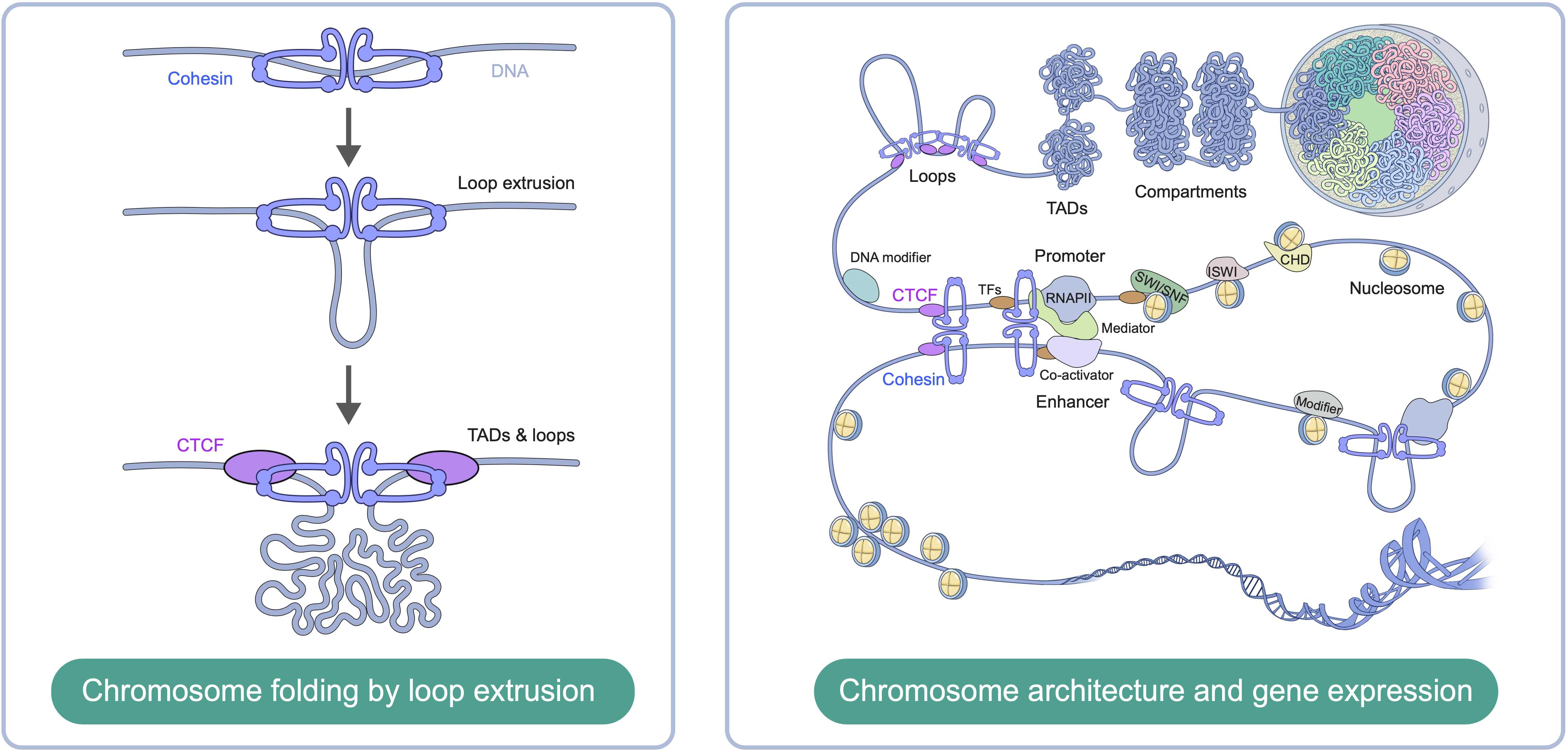
染色体三维结构调控DNA复制、重组与修复,基因转录以及染色体分离等关键细胞活动,并参与细胞分化和个体发育等重要生物学过程。人类基因组DNA总长度约为两米,然而细胞核的直径只有十微米左右。如何将如此长的基因组DNA包装入微米尺度的细胞核中,同时不影响其生物学功能的发挥,这是生命科学悬而未解的基本问题之一。广泛存在于原核和真核细胞中、功能高度保守的染色体结构维持(SMC)复合物在染色体高级结构形成中发挥着至关重要的作用。其中,黏连蛋白(Cohesin)复合物介导哺乳动物细胞分裂间期染色质结构域的形成以及所有真核细胞分裂期姐妹染色单体黏连。在前期工作中,史竹兵博士及其合作者揭示了黏连蛋白激活以及DNA装载的分子机制,并且证实了黏连蛋白发挥染色质压缩功能的“环挤压”模型,为深入理解黏连蛋白调控染色体组织的功能机理奠定了基础(Science,2019;Science,2020;Mol Cell,2023)。
染色体构象与基因表达之间密切联系,相互调控。染色体结构域为基因转录创造了一个微环境,以利于转录的正确且有效进行;而基因转录又可以重塑染色体三维结构,影响染色质环形成以及结构域内相互作用。此外,先锋转录因子结合、表观遗传修饰以及ATP水解驱动的染色质重塑均参与染色体三维结构形成和基因表达调控。史竹兵博士实验室结合生物化学、结构生物学、生物物理学和细胞生物学的方法,探究参与这些生物学过程的大分子机器的三维结构与功能基础,重点关注它们之间的互作机制,从而解决细胞生物学中的基本问题。染色体组织的失调和基因转录的异常是发育障碍疾病以及癌症发生的重要原因。这些研究也将为相关疾病发生机理的阐明提供理论依据。
目前,实验室主要研究内容包括:
1)SMC复合物介导染色体三维结构形成及其调控基因转录分子机制
2)表观遗传修饰包括组蛋白、DNA和RNA修饰及其调控染色体结构功能机理
3)先锋转录因子与染色质重塑复合物之间协同作用基础
4)揭示相关发育障碍疾病和癌症发病原因并寻找针对新靶点的诊治策略

代表论文
(* equal contribution)
1. Zhang H*, Shi Z*, Banigan EJ, Kim Y, Yu H, Bai X, Finkelstein IJ (2023). CTCF and R-loops are boundaries of cohesin-mediated DNA looping. Mol Cell 83(16): 2856-2871. (Preview in Mol Cell: Lifting the curtain on loop extrusion barriers: Single-molecule insights into cohesin stalling)
2. Shi Z, Gao H, Bai XC, Yu H (2020). Cryo-EM structure of the human cohesin–NIPBL–DNA complex. Science 368(6498): 1454-1459. (H1 recommended)
3. Kim Y*, Shi Z*, Zhang H, Finkelstein IJ, Yu H (2019). Human cohesin compacts DNA by loop extrusion. Science 366(6471): 1345-1349. (H1 recommended)
4. Shi Z*, He F*, Chen M, Hua L, Wang W, Jiao S, Zhou Z (2017). DNA-binding mechanism of the Hippo pathway transcription factor TEAD4. Oncogene 36(30): 4362-4369.
5. Shi Z, Jiao S, Zhou Z (2016). STRIPAK complexes in cell signaling and cancer. Oncogene 35(35): 4549-4557. (Review)
6. Hao Q*, Jiao S*, Shi Z*, Li C, Meng X, Zhang Z, Wang Y, Song X, Wang W, Zhang R, Zhao Y, Wong CC, Zhou Z (2015). A non-canonical role of the p97 complex in RIG-I antiviral signaling. EMBO J 34(23): 2903-2920.
7. Jiao S*, Wang H*, Shi Z*, Dong A, Zhang W, Song X, He F, Wang Y, Zhang Z, Wang W, Wang X, Guo T, Li P, Zhao Y, Ji H, Zhang L, Zhou Z (2014). A peptide mimicking VGLL4 function acts as a YAP antagonist therapy against gastric cancer. Cancer Cell 25(2): 166-180.
8. Zhang M*, Dong L*, Shi Z*, Jiao S*, Zhang Z, Zhang W, Liu G, Chen C, Feng M, Hao Q, Wang W, Yin M, Zhao Y, Zhang L, Zhou Z (2013). Structural mechanism of CCM3 heterodimerization with GCKIII kinases. Structure 21(4): 680-688.
9. Shi Z*, Jiao S*, Zhang Z*, Ma M, Zhang Z, Chen C, Wang K, Wang H, Wang W, Zhang L, Zhao Y, Zhou Z (2013). Structure of the MST4 in complex with MO25 provides insights into its activation mechanism. Structure 21(3): 449-461.
10. Wang W*, Shi Z*, Jiao S*, Chen C, Wang H, Liu G, Wang Q, Zhao Y, Greene MI, Zhou Z (2012). Structural insights into SUN-KASH complexes across the nuclear envelope. Cell Res 22(10): 1440-1452.
完整论文列表:https://scholar.google.com/citations?hl=en&user=qNpEU08AAAAJ
联系方式
电子邮箱:shizhubing@westlake.edu.cn